Des biocapteurs à base d’ADN plus performants
Des scientifiques du CNRS décryptent le fonctionnement de bio-nanocapteurs à base d’ADN capables de réaliser des milliers d’analyses génétiques en parallèle. Des résultats, parus dans le Journal of the American Chemical Society, qui devraient permettre d’encore améliorer les propriétés de ces dispositifs en terme de sensibilité par exemple.
Au cours de ces dix dernières années, les biocapteurs à base d’ADN ont envahi le marché à cause de leur faible coût, de leur biocompatibilité, mais surtout grâce à leur miniaturisation extrême qui permet la détection d’une molécule unique. Ces puces d’ADN sont capables de réaliser des milliers d’analyses génétiques en parallèle que ce soit dans le domaine du diagnostic, du génotypage, de la pharmacogénomique ou de l’analyse globale de l’expression des gènes. Les applications de tels dispositifs sont multiples : de la recherche fondamentale jusqu’à la médecine légale (empreinte ADN) aux applications militaires (détection de menaces bactériologiques), en passant par la découverte de nouveaux médicaments et de nouvelles cibles thérapeutiques, le diagnostic de mutations responsables de maladies génétiques…
Typiquement la lecture des puces à ADN est basée sur l’émission de lumière fluorescence par les brins d’ADN et requière un appareillage optique difficilement miniaturisable et transportable. Aussi les chercheurs cherchent-ils à y substituer une lecture électrochimique, plus simple, aisée à miniaturiser, compatible avec les liquides biologiques même opaques, mais jusqu’ici incapable d’atteindre la sensibilité de la molécule unique. La détection électrochimique repose sur la transcription des interactions des brins d’ADN avec leur environnement en un signal mesurable comme le courant électrique. Les dispositifs de ce type sont ainsi capables de détecter d’infimes variations physicochimiques d’espèces moléculaires avec lesquelles ils sont en contact.
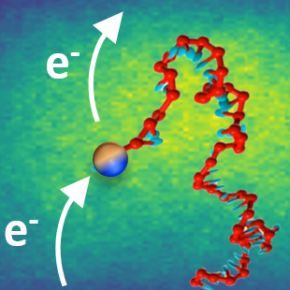
Mais réaliser de telles mesures n’est pas simple. Quelques molécules d'ADN doivent être confinées entre deux électrodes séparées d’une dizaine de nanomètres pouvoir les interroger électrochimiquement et mesurer le signal électrique. Une modification chimique préalable de l’ADN est également nécessaire. On doit en effet greffer aux extrémités des brins des molécules dites « électro actives » ou « rédox » (capables de prendre ou de donner un électron) qui vont transporter les électrons entre les deux électrodes.
Les scientifiques du Laboratoire d’électrochimie moléculaire (CNRS/Université Paris Cité) et du Laboratoire d'analyse et d'architecture des systèmes (LIMMS/LAAS CNRS) viennent de parvenir à immobiliser quelques molécules d'ADN à marqueur redox entre deux électrodes. Pour cela, le brin d’ADN est attaché à une électrode, la seconde électrode pouvant être approchée à quelques dizaines de nanomètres de l’autre extrémité qui porte la molécule redox greffée. La réponse en courant des brins d’ADN a été mesurée et modélisée par des simulations de dynamique moléculaire. Les scientifiques ont ainsi pu caractériser la transmission du courant électrique de la tête redox du brin vers l’électrodes, et montrer que le transport des électrons entre les deux électrodes ne nécessitait pas d’apport énergétique autre que le mouvement Brownien des ADN confinées.
Ces travaux, publiés dans le J. Am. Chem. Soc., ouvrent la voie à la mise au point de biocapteurs à base de brins d’ADN à tête rédox, à priori sensibles à une molécule unique. Le mécanisme de transport d’électron qu’ils ont décrypté, à la fois efficace et peu énergivore, pourrait ne pas être réservé à l’ADN. Son généralisation à toute molécule redox attachée à l’extrémité d’une chaine polymère linéaire nanométrique permettrait aussi d’envisager la conception de nanosystèmes électrocatalytiques à faible consommation d’énergie.
Rédacteur : CCdM
Référence
Zhiyong Zheng, Simon Grall, Soo Hyeon Kim, Arnaud Chovin, Nicolas Clement & Christophe Demaille Activationless Electron Transfer of Redox-DNA in Electrochemical Nanogaps
J. Am. Chem. Soc. 2024
Laboratoire de la circonscription impliqué
Laboratoire d’électrochimie moléculaire (CNRS/Université Paris Cité)