Cartographie des points chauds de mutations du génome d’Escherichia coli
Les erreurs de réplication de l'ADN constituent la principale source de mutations. Savoir où et comment ces erreurs se produisent est crucial pour comprendre les mécanismes de la stabilité du génome. Dans un article publié dans Nucleic Acids Research, des scientifiques ont réalisé une cartographie exhaustive des points chauds d’erreurs de réplication chez la bactérie Escherichia coli. Ils montrent que ces points chauds ne sont pas disposés de manière aléatoire sur le génome de la bactérie.
Les mutations peuvent être délétères pour les organismes, mais elles constituent également le moteur de l’évolution en permettant l’adaptation. La principale source de mutations provient des erreurs de réplication de l’ADN, qui surviennent malgré les multiples mécanismes de contrôle, et de réparation. Identifier précisément où et comment ces erreurs se produisent est crucial pour comprendre les mécanismes de stabilité génomique.
Une technique innovante pour localiser les erreurs de réplication.
Dans une étude publiée dans la revue Nucleic Acids Research, les scientifiques ont utilisé une stratégie innovante en exploitant le système de réparation des mésappariements chez Escherichia coli. Ils ont modifié ce système pour qu’il puisse détecter les erreurs sans les corriger. En marquant la protéine de réparation des mésappariements MutL avec une protéine fluorescente, ils ont ainsi pu visualiser les erreurs de réplication de l’ADN au fur et à mesure de leur apparition dans des cellules vivantes. Grâce à une technique appelée MutL-ChIP-seq, qui permet d’analyser les interactions entre protéines et ADN, ils ont aussi identifié des régions génomiques propices aux erreurs de réplication.
Des régions du génome sous haute pression.
Les résultats montrent que les points chauds d’erreurs de réplication ne sont pas distribués de manière aléatoire, mais sont associés à des régions génomiques particulières :
- Des séquences thermiquement instables facilitant la séparation des brins d’ADN ;
- Des répétitions mononucléotidiques favorisant des glissements de l’ADN polymérase ;
- Des structures secondaires, comme les structures cruciformes, susceptibles de bloquer la progression de la réplication.
Ces points chauds présentent également une forte concentration de sites de liaison pour les protéines impliquées dans la structuration du génome et la régulation de la transcription, ainsi qu’une activité transcriptionnelle élevée.
En conclusion, cette étude fournit une cartographie exhaustive des points chauds d’erreurs de réplication dans le génome bactérien soulignant les interactions complexes entre les facteurs topologiques, structuraux et transcriptionnels qui influencent la fidélité de la réplication. Ces découvertes ouvrent des voies prometteuses pour mieux comprendre les mécanismes menant aux mutations et à l'instabilité génétique.
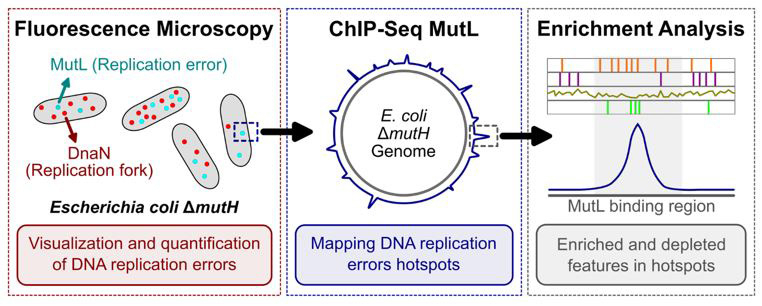
Figure : Visualisation des erreurs de réplication de l'ADN et identification des points chauds d'erreurs de réplication. Panneau de gauche : Les erreurs de réplication de l'ADN sont détectées et quantifiées à l'aide de protéines marquées par fluorescence : MutL (cyan), qui marque les erreurs de réplication, et DnaN (rouge), qui marque les fourches de réplication. Cela permet de cartographier précisément les erreurs de réplication et les fourches de réplication dans les cellules d'E. coli incapables de réparer les erreurs de réplication. Panneau du milieu : Comme les erreurs de réplication ne sont pas corrigées, nous avons réalisé une analyse MutL-ChIP-Seq pour identifier les points chauds d'erreurs de réplication dans le génome. Panneau de droite : L'analyse détaillée des régions de liaison de MutL a révélé des caractéristiques génomiques enrichies ou appauvries dans ces points chauds, fournissant une carte complète à l'échelle du génome des points chauds d'erreurs de réplication et des informations sur les déterminants moléculaires de la fidélité de la réplication.
En savoir plus : Hasenauer, F.C., Barreto, H.C., Lotton, C., and Matic, I. (2024) Genome-wide mapping of spontaneous DNA replication error-hotspots using mismatch repair proteins in rapidly proliferating Escherichia coli. Nucleic Acids Res. https://doi.org/10.1093/nar/gkae1196
Laboratoire
Institut Cochin (CNRS/Inserm/Université Paris Cité)
22 rue Méchain,
75014 Paris

